Des archées pour mieux comprendre le cancer

Récemment on a caractérisé un troisième domaine du vivant, les archées, aux propriétés intermédiaires entre les organismes de type humain ou végétal (qui possèdent des cellules avec noyaux) et les bactéries. Elles sont des championnes de l’adaptation et de la stabilité génomique. Ainsi, des essais de modification de leur génome par l’action de certaines protéines ont permis des avancées sur le rôle de celles-ci dans la prédisposition au cancer.
Dans les cellules de patients atteints de maladies génétiques rares dites « maladies cassantes » (e.g. anémie de Fanconi, syndrome de Bloom…), un défaut des mécanismes de surveillance et de réparation des lésions de l’ADN provoque des cassures de chromosomes.
L’étude de ces maladies a permis d’établir un lien entre instabilité génétique, c’est-à-dire l’altération physique de la molécule d’ADN au sein des cellules, et la prédisposition au cancer. Ainsi, comprendre les mécanismes moléculaires permettant d’assurer la stabilité du génome constitue un enjeu majeur.
Le modèle des bactéries
Au sein de ce que l’on appelle le monde du vivant, nous, humains, faisons partie des eucaryotes, domaine du vivant qui regroupe les animaux et les végétaux, et correspond aux organismes dont l’ADN est dans un compartiment spécifique de la cellule qu’on appelle le noyau.
Un deuxième domaine du vivant est constitué des bactéries, microorganismes dits procaryotes dont l’ADN n’est pas dans un compartiment spécifique.
Cependant, les principes fondamentaux de la stabilité du génome étant conservés chez tous les êtres vivants, l’utilisation des bactéries comme organismes modèles a permis de comprendre les bases moléculaires de l’instabilité génétique, et d’en faciliter l’étude chez des organismes plus complexes, dont l’homme.
Les riches possibilités des archées
Depuis les années 1970, un troisième domaine du vivant a été identifié, qu’on appelle les archées. Ce sont des microorganismes procaryotes, longtemps assimilés à des bactéries du fait de leur ressemblance morphologique.
C’est leur caractérisation au niveau moléculaire, par Carl Woese et George E. Fox, qui a montré que ces microorganismes sont aussi différents des bactéries qu’ils le sont des eucaryotes 1.
Leur caractérisation a également montré que les protéines intervenant dans les processus de réplication et de réparation de l’ADN ressemblent à celles rencontrées chez les eucaryotes. Par ailleurs, une autre caractéristique remarquable des archées est leur incroyable capacité à coloniser des environnements extrêmes.
Certaines archées sont retrouvées au niveau de cheminées hydrothermales océaniques, comme Pyrococcus abyssi (103 °C, pression de 200 atmosphères). D’autres colonisent les sources chaudes volcaniques, dont Sulfolobus acidocaldarius, capable de se développer dans des eaux au pH acide (55 °C‑90 °C, pH 1–5).
Des champions de l’adaptation et de la stabilité génomique
Il est important de préciser que les archées sont également présentes dans de nombreux environnements comme le sol, les océans, la flore intestinale, ou encore… notre nombril.
La similarité de leurs protéines du métabolisme de l’ADN avec celles des eucaryotes, et l’adaptation de ces métabolismes à des conditions de vie extrêmes, font des archées des modèles d’études originaux et pertinents que nous avons choisi d’étudier pour apporter des connaissances fondamentales sur les mécanismes moléculaires qui garantissent la stabilité du génome.
Nous utilisons pour cela Haloferax volcanii, archée halophile (adaptée à de fortes concentrations en sels) que l’on retrouve dans la mer Morte et les grands lacs salés. Elle présente l’avantage d’être facilement manipulable en laboratoire par des outils génétiques permettant de modifier son génome pour étudier le rôle des protéines au sein de la cellule vivante.
Le rôle clef de la protéine Hef
Nous nous intéressons au rôle de la protéine Hef, qui possède deux activités enzymatiques : une activité hélicase d’ouverture de l’ADN double brin, et une activité nucléase de coupure de l’ADN. On la retrouve uniquement chez les archées et les eucaryotes.
Plusieurs protéines de cette famille sont retrouvées chez l’homme, dont certaines sont impliquées dans des maladies génétiques entraînant des prédispositions au développement de cancer, et dont le rôle n’est pas encore complètement compris. C’est notamment le cas de la protéine FANCM, impliquée dans l’anémie de Fanconi.
Nous avons réalisé une étude génétique, qui consiste à observer les défauts qu’entraîne l’absence de la protéine dans la cellule. Cela nous a permis de proposer que Hef agisse lors d’arrêt de la réplication, pour permettre le redémarrage 2.
En effet, la réplication de l’ADN permet à toute cellule qui prolifère de dupliquer son matériel génétique. Ainsi, en cas d’arrêt de la réplication, son redémarrage est essentiel pour la survie.
Nous avons également mis en évidence une voie alternative impliquant une autre nucléase, appelée Hjc. Seule l’absence des deux protéines simultanément ne permet plus le redémarrage, les arrêts de réplication conduisant à la mort cellulaire.
Des analyses concluantes menées avec le LOB
Pour aller plus loin dans la compréhension du rôle de Hef, en collaboration avec nos collègues « microscopistes » du Laboratoire d’optique et biosciences (LOB), nous nous sommes tournés vers la microscopie de fluorescence couplée à de l’analyse d’images quantitative dans le but de localiser Hef dans la cellule vivante.
Pour la visualiser, grâce à notre expertise en biologie moléculaire et en génétique, nous avons exprimé Hef fusionnée à une protéine qui possède l’exceptionnelle propriété d’être intrinsèquement fluorescente, appelée protéine verte fluorescente (GFP). Nous avons pu observer que Hef forme des foyers de fluorescence dans les cellules, dont le nombre augmente considérablement lorsque l’on inhibe la synthèse de l’ADN par l’ajout d’une drogue, provoquant un stress réplicatif.
L’ensemble de nos résultats a permis de montrer que Hef est recrutée aux sites d’arrêt de la réplication, validant le modèle proposé lors de l’étude génétique 3, 4.
Comprendre le dynamisme et l’arrêt de réplication de l’ADN
Pour mieux appréhender la dynamique de réplication au sein d’Haloferax volcanii, et notamment comprendre comment est orchestré le rôle de Hef lors des arrêts de réplication, nous nous intéressons également aux protéines du réplisome, complexe protéique coordonnant les différentes activités enzymatiques nécessaires à la synthèse de l’ADN.
Dans le but d’étudier la dynamique de la réplication au sein des cellules vivantes, nous avons fusionné RPA2, protéine de fixation à l’ADN simple brin, à la GFP. RPA2 présente une localisation très dynamique, et reflète des foyers de réplication actifs en conditions normales, des foyers où la réplication est arrêtée en réponse à un stress réplicatif (comme c’est le cas pour Hef) et des foyers de réparation de l’ADN en réponse à des dommages de l’ADN.
La résolution de ces derniers a nécessité l’utilisation d’une technique de microscopie de superrésolution par illumination structurée, grâce à l’acquisition de cet équipement par la plateforme d’imagerie morphoscope développée au laboratoire dans le cadre des équipements d’excellence financés par les investissements d’avenir du ministère de l’Enseignement supérieur, de la Recherche et de l’Innovation.
Des belles perspectives de recherche
Cette recherche interdisciplinaire nous a permis de montrer que Hef a effectivement un rôle très important dans le redémarrage des fourches de réplication, et de développer des outils puissants qui nous ouvrent de larges perspectives pour comprendre les bases moléculaires de la stabilité du génome chez les archées.
BIBLIOGRAPHIE
- “Phylogenetic structure of the prokaryotic domain : the primary kingdoms”. Proc Natl Acad Sci USA vol.74 (11): 5088–5090.
- Lestini, R., Laptenok, S.P., Kuhn, J., Hink, M.A., Schanne- Klein, M.C., Liebl, U. and Myllykallio, H. (2013) “Intracellular dynamics of archaeal FANCM homologue Hef in response to halted DNA replication”. Nucleic Acids Res, 41, 10358–10370.
- Lestini, R., Duan, Z. and Allers, T. (2010) “The archaeal Xpf/Mus81/FANCM homolog Hef and the Holliday junction resolvase Hjc define alternative pathways that are essential for cell viability in Haloferax volcanii”. DNA Repair (Amst), 9, 994‑1002.
- Lestini, R., Delpech, F. and Myllykallio, H. (2015) “DNA replication restart and cellular dynamics of Hef helicase/ nuclease protein in Haloferax volcanii”. Biochimie, vol. 118, 254–263.
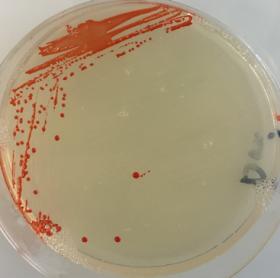
En illustration : Colonies d’Haloferax volcanii sur boîte de Petri. Les membranes de ces cellules sont riches en pigments rouges qui les protègent du rayonnement UV et sont responsables de la couleur rouge des lacs salés.