Les tours de passe-passe des topoisomérases
Le contexte biologique
Le contexte biologique
Il y a cinquante ans, Crick et Watson décrivaient la structure de la double hélice et faisaient remarquer que l’existence des deux brins complémentaires permettait de proposer un mécanisme de réplication de la molécule dans lequel il suffisait de séparer les deux brins et de recopier chacun en formant son complémentaire pour obtenir enfin deux molécules d’ADN filles rigoureusement identiques à la molécule parente. Cette intuition devint une réalité dans les années 1970–1980 lorsque les enzymes chargées du recopiage, les polymérases, ainsi que celles chargées de séparer les brins complémentaires, les hélicases, furent identifiées puis isolées.
La structure de la double hélice implique que les deux brins d’ADN complémentaires s’entortillent l’un autour de l’autre. Par ailleurs, les molécules d’ADN sont très longues (~10 cm pour un chromosome) et l’on compte un tour d’hélice tous les 3,4 nanomètres. En conséquence, le nombre de tours impliqué est gigantesque. Or, le bon fonctionnement du mécanisme de duplication de l’ADN implique que tous les tours, jusqu’au dernier, soient débobinés afin de pouvoir séparer les deux molécules filles. Afin d’accommoder cette contrainte majeure et très délicate, on imaginait simplement que les molécules tournaient au niveau de leurs extrémités. De plus, la complexité de ce problème s’est encore accrue dans le cas des bactéries : dans ces organismes, l’ADN adopte la forme d’un anneau fermé par des liaisons chimiques covalentes. En conséquence, les deux brins de la double hélice sont topologiquement inséparables. Dix ans après la découverte de Crick et Watson, ce problème a même poussé certains à dire que la double hélice n’était probablement pas la bonne structure !
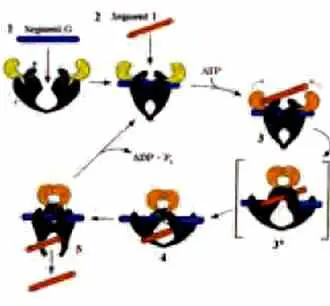
C’est James Wang qui dans les années soixante-dix apporta la solution à l’énigme [1]. Il découvrit une nouvelle classe d’enzyme, les topoisomérases, qui sont capables de changer la topologie de la molécule d’ADN en effectuant une coupure temporaire dans la molécule pour y faire passer soit un brin, soit les deux brins de la double hélice. Ce faisant, ces enzymes permettent de relâcher les contraintes de torsion sur une molécule ou de désenchevêtrer deux molécules entortillées.
La découverte des topoisomérases a permis de résoudre le problème des nœuds dans les molécules d’ADN. Cependant, leur fonctionnement a soulevé d’autres questions : comment des enzymes mesurant quelques nanomètres peuvent-elles relâcher jusqu’au dernier tour d’entortillement des molécules qui s’étendent sur des distances jusqu’à un million de fois plus grandes ? Comment ces enzymes déterminent-elles de quel côté de la brèche il faut transférer une molécule afin de défaire un nœud et non pas, au contraire, en créer de nouveaux ? Les réponses à ces questions nous manquent encore à l’heure actuelle. Certains de nos résultats suggèrent que ces enzymes reconnaissent l’angle formé par les molécules lors de leur croisement. En particulier, un type de topoisomérase que l’on trouve chez les bactéries se comporte très différemment selon l’angle de croisement des molécules Cependant, les vérifications expérimentales de ces hypothèses sont particulièrement délicates à réaliser dans des expériences faites en tube à essai. En effet, dans ce contexte, comme les molécules d’ADN sont soumises au mouvement brownien, l’angle qu’elles adoptent lors de leur croisement est largement aléatoire.
Néanmoins, depuis quelques années, des expériences peuvent être réalisées à l’échelle d’une seule molécule. Ces expériences permettent de contrôler les paramètres physiques d’une molécule et d’imposer, comme nous le décrivons ci-dessous, l’angle de croisement entre deux molécules.
La micromanipulation par pinces magnétiques
Ce sont les groupes de S. Chu et C. Bustamante qui ont réalisé les premières expériences de micromanipulation de molécules uniques [2]. Celles-ci se font à l’aide d’un microscope optique et dans le milieu naturel de la molécule d’ADN, c’est-à-dire de l’eau avec un peu de sel. Dans ces conditions, l’observation directe de la molécule est impossible.
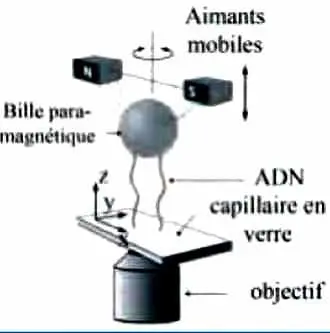
Par contre, en utilisant un « scotch moléculaire » il est assez facile d’attacher une bille de quelques microns à une extrémité de la molécule et de visualiser ainsi ses mouvements. Mieux, on peut attacher de façon analogue la seconde extrémité de la molécule à la paroi du récipient. En exerçant une force sur la bille, on peut ainsi étirer la molécule d’ADN. Le « scotch moléculaire » est un couple de molécules de type « clef-serrure » : la clef est une petite molécule (de la biotine par exemple) qui possède une affinité très importante pour une molécule plus grosse qui en épouse la forme (de la streptavidine dans le cas de la biotine). La molécule d’ADN est préparée en attachant chimiquement la biotine à une extrémité. Les billes magnétiques sont recouvertes de streptavidine.
En plaçant les molécules d’ADN ainsi préparées en présence des billes en solution, celles-ci se couplent spontanément aux billes de façon quasi irréversible. L’accrochage de la seconde extrémité de la molécule se fait par un deuxième jeu clef-serrure (digoxigénine-antidigoxigénine). Comme l’accrochage des billes aux molécules d’ADN se fait par diffusion, rien n’empêche deux ou plusieurs molécules d’ADN de relier la bille à la surface du récipient. En pratique, nous ajustons la concentration relative des molécules aux billes pour que la plupart de celles-ci soient reliées par une seule molécule d’ADN. (Les billes qui ne sont attachées à aucune molécule sont éliminées par rinçage.) Cependant, statistiquement un petit nombre de billes se trouvent être attachées par deux molécules d’ADN, cette configuration va nous permettre de croiser à volonté deux molécules.
Au cours de leur mouvement brownien, les molécules d’eau bousculent l’ADN dans tous les sens et tendent à lui faire adopter la forme d’une pelote fluctuante. Il faut donc appliquer une force pour étirer la molécule ; ceci se fait en agissant sur la bille micrométrique qui localise une extrémité de la molécule. Il existe plusieurs moyens pour appliquer cette force. D’abord, les pinces optiques, qui utilisent un faisceau laser convergeant qui attire la bille près de son point de focalisation. Ensuite, les pinces magnétiques, basées sur l’utilisation d’aimants qui attirent la bille contenant un matériau magnétique. Cette seconde méthode permet également de faire tourner la bille simplement en faisant tourner les aimants.
Dans le cas des pinces magnétiques, pour une position donnée des aimants par rapport à la bille, la force appliquée est constante dans le domaine que peut explorer la bille. Pour déterminer cette force, il suffit de mesurer l’ampleur du mouvement brownien de la bille. Pour les faibles forces ces mouvements sont importants ; plus on rapproche les aimants, plus la force de traction augmente et plus l’ampleur du mouvement brownien diminue. La bille attachée à la molécule d’ADN sous l’action des aimants se comporte comme un pendule inversé, la force magnétique la tirant vers le haut.
En appliquant le théorème de l’équipartition, on montre que F = l.kBT/< x2 > [3] (où l est l’extension de la molécule, < x2 > est l’amplitude quadratique moyenne du mouvement brownien, kB la constante de Boltzmann et T la température absolue). La force typique qu’il faut appliquer pour étirer une molécule d’ADN est de l’ordre de 1 pN (10-12 N), mille fois plus faible que la force de rupture de l’ADN (~1 000 pN).
Pour mesurer le mouvement brownien, nous avons écrit un programme de traitement d’images vidéo qui suit la position horizontale de la bille en temps réel, avec une précision de quelques nanomètres. En analysant les motifs de diffraction de l’image de la bille obtenue en éclairage parallèle, il est également possible d’obtenir la position verticale de la bille avec une précision comparable. Cette mesure nous permet ainsi de déterminer la distance séparant la bille de la paroi du récipient.
Le vrillage d’une balançoire
| FIGURE 3 — Tours de passe-passe moléculaire |
 |
|
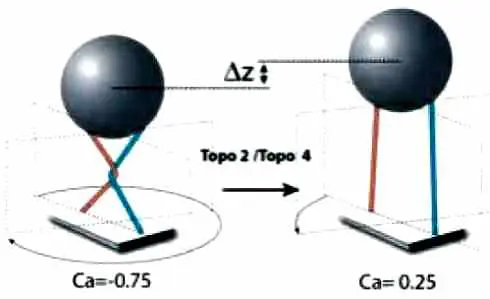
Principe d’action de la topoisomérase sur le croisement de deux molécules d’ADN. En faisant effectuer un tour à la bille avec les aimants, nous pouvons passer d’une situation où les molécules ne présentent pas de croisement (droite) à celle où elles se croisent (gauche). La topoisomérase reconnaît alors le croisement, et en opérant l’action décrite dans la figure 1, l’enzyme dénoue le croisement et ramène les deux molécules dans la situation sans croisement (droite). Dès lors, la contrainte étant relâchée, l’enzyme ne peut plus agir. Le changement de hauteur entre les deux configurations permet de déterminer le moment où l’action de l’enzyme s’effectue. Par ailleurs, dans l’hypothèse où les points d’attache des molécules sont séparés par des distances équivalentes, le changement relatif de hauteur entre les deux configurations est égal à (1 – cosq/2) où q est l’angle de croisement des deux molécules. Si nous ajoutons maintenant des topoisomérases dans la solution avec un peu d’ATP (la source d’énergie nécessaire à la plupart des opérations enzymatiques), il ne se passe rien si les deux molécules sont parallèles. Par contre, si nous créons un point de croisement en imprimant un tour à la bille, l’extension de la molécule diminue pour les raisons décrites ci-dessus. Alors, une topoisomérase va s’accrocher au croisement et le supprimer, permettant à l’extension de reprendre sa valeur maximale de départ. On peut alors imprimer un nouveau tour à la bille générant un nouveau croisement que l’enzyme va s’empresser de dénouer, etc. Chaque événement correspond à un seul cycle enzymatique d’autant plus facile à détecter que le changement d’extension correspond à une fraction de micron comme on peut le voir sur la figure 4. |
Il est assez facile de sélectionner les billes ancrées à la paroi par deux molécules : ces molécules sont typiquement séparées par une distance comparable au rayon de la bille. En faisant tourner celle-ci d’un demi-tour dans un sens ou dans l’autre, les deux molécules sont amenées à se croiser. Si la longueur des molécules est comparable au diamètre de la bille, il se produit alors un raccourcissement notable de la distance séparant la bille à la paroi, comme on peut l’observer en faisant vriller une balançoire autour de son axe.
Cette variation rapide d’extension sur un tour n’est visible que sur des billes accrochées par deux molécules. Ces billes nous fournissent un moyen simple de croiser deux molécules avec un angle donné que nous évaluons en mesurant le raccourcissement provoqué par un demi-tour comparé à la longueur des molécules d’ADN dans leur configuration parallèle. Évidemment, pour une longueur donnée de la molécule d’ADN, cet angle dépend de la distance séparant les deux molécules que nous ne contrôlons pas. Cependant en allant à la pêche aux billes, on peut réaliser un échantillonnage de différents angles de croisement s’étalant de 50° à plus de 100° [4].
Comme illustré en figure 5, le point de croisement de deux molécules chirales d’ADN formant un angle n’est pas le symétrique de la configuration correspondant à l’angle – θ. Par contre, les situations – θ et π – θ sont, elles, symétriques [6]. Dans notre expérience, en tournant la bille d’un demi-tour dans le sens des aiguilles d’une montre nous obtenons un angle de croisement θ, en tournant dans l’autre sens, nous obtenons – θ. En employant la rotation des aimants pour générer le substrat topologique (c’est-à-dire l’angle) voulu, il est aisé de mesurer le temps mis par la topoisomérase IV pour dénouer le croisement correspondant à θ et – θ. La valeur de l’angle θ est déterminée à partir du changement de hauteur de la bille et de la longueur des molécules.
Pour les petits angles de croisement (correspondant à des billes dont la hauteur change peu en passant de la configuration molécules parallèles à molécules croisées), nous observons que le temps d’action moyen de la topoisomérase IV est vingt fois plus long pour la configuration à 50° que celle correspondant à – 50°. Pour les billes qui présentent une variation de hauteur importante, l’angle de croisement approche 90° et peut même dépasser cette valeur. Or comme nous l’avons expliqué la situation de croisement à 90° est identique à celle de croisement à – 90°. Ainsi nos expériences montrent que pour un angle de θ = + 76° ou – 76° les temps moyens d’action ne diffèrent que de 10 %.
Puisque les molécules d’ADN sont animées de fluctuations browniennes importantes, leur angle de croisement présente en fait une distribution et la valeur de l’angle de croisement dont nous avons parlé est en fait la valeur moyenne. La largeur de cette distribution dépend de la force de traction appliquée aux molécules. Si la force est très grande, les molécules sont quasiment rectilignes et la distribution des angles est étroite ; aux faibles forces c’est l’inverse. Dans notre expérience, nous observons bien que la sélectivité angulaire est renforcée avec la force appliquée sur la bille.
La topoisomérase IV est très habile lors de son tour de passe-passe moléculaire, jamais nous ne l’avons surprise à lâcher les brins coupés avant de les recoller (la bille se retrouverait alors accrochée par une seule molécule). Un tel accident serait dramatique au sein de nos chromosomes : il conduirait à une cassure double brin qui peut certes être réparée par des mécanismes cellulaires adaptés mais avec un taux d’échec très gênant.
| FIGURE 4 |
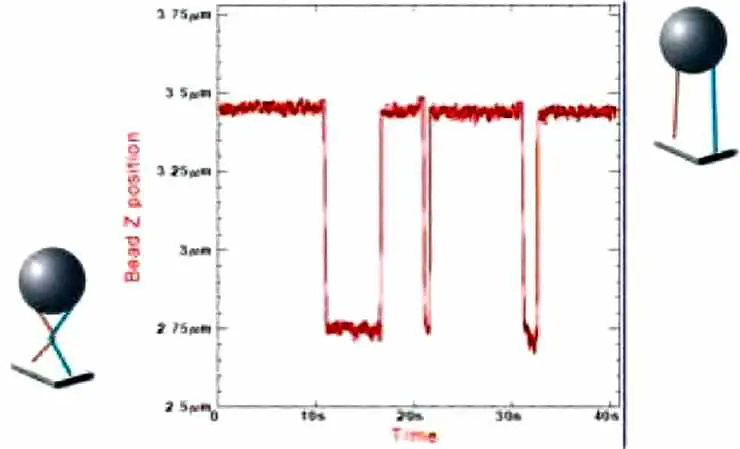 |
|
Signal expérimental permettant de voir l’action de la topoisomérase : le graphique représente la distance entre la bille et la paroi de verre. Lorsque celle-ci atteint la valeur de 3.45 microns, les deux molécules sont parallèles. Quand les molécules se croisent la distance se réduit à 2.75 microns. En présence de topoisomérases dans la solution on n’observe aucun changement de longueur lorsque les molécules sont parallèles. Par contre chaque fois que l’on fait faire un tour aux aimants, dans un premier temps, la bille se rapproche de la paroi. Au moment où l’enzyme dénoue le croisement, on observe une remontée brutale de l’extension. Sur cet enregistrement, nous avons répété l’opération trois fois, à chaque fois l’enzyme a agi, cependant elle l’a fait après un temps très variable. Le temps mis par l’enzyme pour libérer le croisement après sa formation correspond au temps de diffusion de l’enzyme pour trouver le point de croisement et au temps de fixation sur ce croisement. Il dépend évidemment de la concentration d’enzyme ; mais d’un cycle au suivant ce temps est une variable aléatoire présentant une distribution statistique de Poisson avec un temps caractéristique t. Pour une concentration enzymatique de l’ordre du nano-molaire, t est typiquement de quelques secondes. Si nous tournons la bille de plusieurs tours rapidement, après un temps d’attente, une enzyme déjà sur place enchaîne une série de cycles avec une cadence de 2 ou 3 à la seconde [5]. |
Conclusion
| FIGURE 5 — Les topoisomérases reconnaissent l’angle de croisement des molécules |
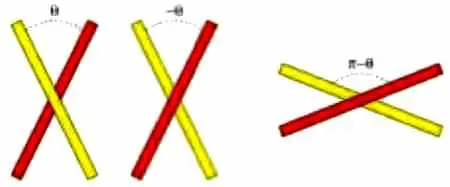 |
|
Symétrie angulaire impliquée dans le croisement de deux molécules d’ADN. La situation correspondant à l’angle ‑q (au centre) est différente de la situation θ (à gauche), en effet ces deux configurations découlent de la symétrie miroir (c’est une situation chirale), par contre la situation correspondant à l’angle -θ (au centre) est équivalente à π – θ (à droite). Notons que pour θ = 90 les différentes configurations sont identiques. |
Nous avons développé des techniques de micromanipulation de molécules uniques. Ces techniques nous ont permis de mettre en évidence les mécanismes précis qu’utilisent certaines enzymes pour déplier et réduire les tensions dans les molécules d’ADN.
Il s’agit d’un sujet de recherche très actif actuellement et plusieurs groupes de recherche ont obtenu des résultats remarquables sur les moteurs moléculaires, les polymérases, les hélicases, etc. Ces résultats viennent naturellement compléter ceux obtenus en tube à essai. Ils démontrent, s’il était nécessaire, que ces enzymes sont de magnifiques machines capables de travailler avec une remarquable précision dans un environnement agité par le mouvement brownien. Les topoisomérases sont pour le moins des enzymes extraordinaires du fait qu’en bien des points elles surpassent ce que nous savons faire à l’échelle macroscopique.
Ainsi il nous reste encore à comprendre comment ces machines de taille nanométrique dénouent fidèlement des molécules mille fois plus grandes qu’elles.
Remerciements
Les expériences décrites ici n’auraient pas été possible sans l’aide de J.-F. ALLEMAND, O. SALEH, H. YOKOTA, T. LIONNET, M. DUGUET et le support financier de l’ENS, du CNRS, des universités Paris VI et VII, de la CEE et de l’ARC.